近日,意大利锡耶纳大学的研究人员在预印本平台 bioRxiv 上发表了题为:Evidence for RNA editing in the transcriptome of 2019 Novel Coronavirus 的研究论文。研究者分析了2名武汉患者肺泡灌洗液中的新型冠状病毒(SARS-CoV-2)的RNA序列发现:人类宿主的脱氨酶(APOBECs/ADARs)参与SARS-CoV-2转录组的RNA编辑,这一过程可能对SARS-CoV-2与患者的相互作用造成深远影响。  病毒是专性细胞内寄生物,在经过漫长的协同进化过程,宿主体内进化出天然免疫系统来感知和抑制病毒的入侵,其中包括内源性脱氨酶介导的DNA和RNA编辑。哺乳动物中存在两类脱氨酶:1、ADARs靶向双链RNA,将腺嘌呤(A)脱氨转换成次黄嘌呤(I);2、APOBECs靶向单链RNA,将胞嘧啶(C)脱氨转换成胸腺嘧啶(U)。 APOBECs/ADARs可以干扰病毒的复制过程,包括冠状病毒,但其酶活性是否参与尚不清楚。为了评估宿主对SARS-CoV-2感染反应中是否发生RNA编辑,研究者从公开数据库中获取了武汉COVID-19患者的肺泡灌洗液(BALF)中分离到的SARS-CoV-2的RNA序列数据集, 测序质量因样本而异,其中只有2个样本具有适合于识别潜在编辑位点的覆盖率和错误率。 研究人员通过REDItools 2对这2个样本数据进行分析,共鉴定了487个单核苷酸变异(SNVs),其中41个来自患者SRR10903401, 446个来自患者SRR10903402。SARS-CoV-2转录组的SNVs鉴定SNVs的等位基因比例在1-5%之间,并且存在转换偏向性,转换次数与冠状病毒的突变率(10-6/-7)一致。研究人员观察到SARS-CoV-2转录组中A→G/T→C的变化,这可能是ADARs介导的A→I造成的;而C→T/G→A的转变,则可能由APOBECs介导的C→U造成的。 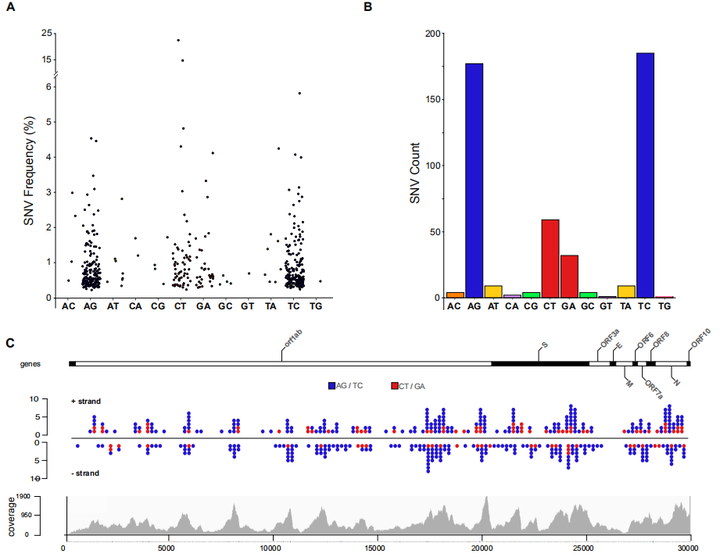 值得注意的是,SNVs在大多数病毒基因上的积累与基因长度和测序深度成正比,唯一的例外是调节干扰素信号的ORF6基因,该基因的编辑率高于其他基因。 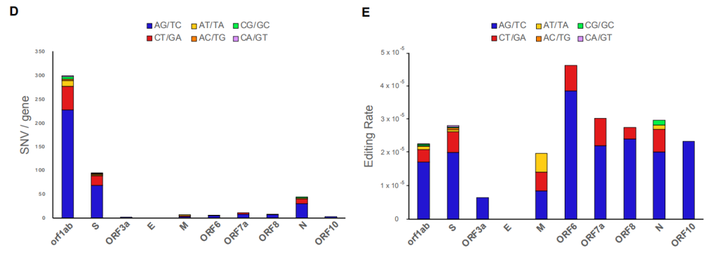 SARS-CoV-2 RNA编辑位点的序列特征由于APOBECs脱氨酶优先在特定的序列环境中靶向胞嘧啶,研究人员分析了SNVs的三核苷酸环境,基因组归一化后,A→I变化没有明显的偏差。C→U的变化主要发生在胸腺嘧啶和腺嘌呤的3 '位上,与APOBEC1脱氨基相兼容的模式([AU]C[AU])相一致。此外,最常被编辑的三核苷酸是TCG,它与APOBEC3A模式(UCN)兼容。  冠状病毒科不同病毒株的核苷酸变化研究人员对SARS-CoV-2、MERS-CoV和SARS-CoV等现有冠状病毒基因组进行比对,以验证RNA编辑是否可能对通过进化获得的某些突变负责。基因组比对显示,所有病毒株中相当一部分突变可能来自脱氨基作用导至的C→U和A→I,并且一种与APOBEC介导的编辑兼容模式也存在于基因组C→U的SNVs中。 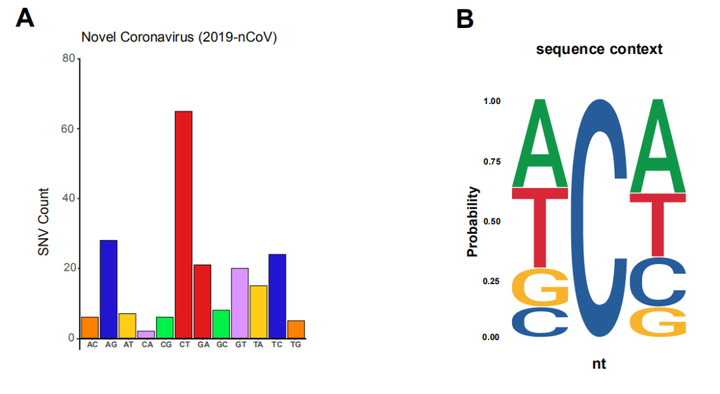 RNA编辑在SARS-CoV-2中的功能意义还有待探究:对入侵病毒基因组的编辑可能导至病毒死亡,也可能促进病毒进化。对RNA病毒而言,对其RNA编辑,造成其死亡的可能性比导至其进化可能性更大,因为这将直接影响它的遗传信息。 文章来源:BioWorld |